Mamot Adam
Zakład Chemii Organicznej i Technologii Chemicznej
Pracownia Syntezy Metaloorganicznej
Synteza i badanie właściwości analogów oligofosforanów 7-metyloguanozyny modyfikowanych w pozycji C8 – potencjalnych inhibitorów białek zaangażowanych w metabolizm mRNA
Adam Mamot
Promotor: Dr hab. Michał Barbasiewicz
Opiekun: Dr hab. Jacek Jemielity, prof. UW
Na końcu 5’ eukariotycznych mRNA znajduje się struktura kapu (ang. 5’ mRNA cap structure), która ze względu na swoją unikalną budowę bierze udział w wielu, kluczowych dla przetrwania komórki, etapach procesu ekspresji genów.[1] Składa się ona z
7-metyloguanozyny połączonej mostkiem 5’-5’ trifosforanowym z pierwszym nukleotydem transkryptu. Analogi struktury kapu wykorzystywane są jako narzędzia w badaniach biochemicznych i biofizycznych oraz jako związki o potencjale terapeutycznym.[1,2] W mojej pracy opisałem syntezę serii mononukleotydowych analogów struktury kapu – oligofosforanów (mono-, di- i trifosforanów) 7-metyloguanozyny, zawierających w pozycji C8 podstawniki fenylowe, metylowe lub cyklopropylowe (Rys.). Do tej pory nie opublikowano badań mówiących o wpływie tego typu modyfikacji na właściwości 7-metyloguanozyny czy jej oddziaływań z białkami wiążącymi kap.
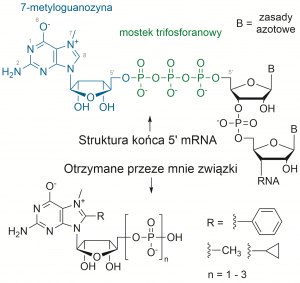
Związki te zawierają jedną, dwie lub trzy ujemnie naładowane grupy fosforanowe, różnicujące je pod względem wypadkowego ładunku i długości łańcucha oligofosforanowego. Ponadto, w obrębie zasady azotowej, zawierają wszystkie kluczowe elementy wymagane do wiązania przez białka rozpoznające 7-metyloguanozynę: dodatnio naładowany układ aromatyczny oraz wolne atomy N1, N2 i O6. Z kolei, różne pod względem rozmiarów i charakteru chemicznego podstawniki w pozycji C8 mogą potencjalnie modulować siłę oddziaływań pomiędzy różnymi białkami wiążącymi 7-metyloguanozynę.
Kluczowym etapem syntezy była reakcja Suzuki-Miyaura, prowadzona na odpowiednim oligofosforanie 8-bromoguanozyny. Pozwala ona na dużą swobodę w doborze wprowadzanych podstawników, co w połączeniu z odpowiednimi badaniami przesiewowymi (ang. screening) zwiększa szansę na odnalezienie selektywnych inhibitorów białek regulujących metabolizm mRNA. Wszystkie produkty końcowe i pośrednie scharakteryzowałem za pomocą dostępnych technik spektroskopowych i chromatograficznych (NMR, UV-vis, MS, HPLC).
To podejście pozwoliło szybko i wydajnie otrzymać wiele nowych związków o nieudokumentowanych do tej pory właściwościach biologicznych. W pracy prezentuję również wyniki wstępnych badań oddziaływań otrzymanych związków z białkami eIF4E, DcpS i cNIIIB. Zastosowanie tej metodologii może pozwolić na lepsze zrozumienie mechanizmów działania czy nawet odnalezienie selektywnych inhibitorów białek wiążących kap.
Literatura:
[1] Warmiński M., Sikorski P. J., Kowalska J., Jemielity J., Top. Curr. Chem. 2017, 375, 16-29.
[2] Mamot A., Warmiński M., Sikorski P. J., Kowalska J., Jemielity J., Angew.Chem.Int. Ed. 2017, 56, 15628–15632.