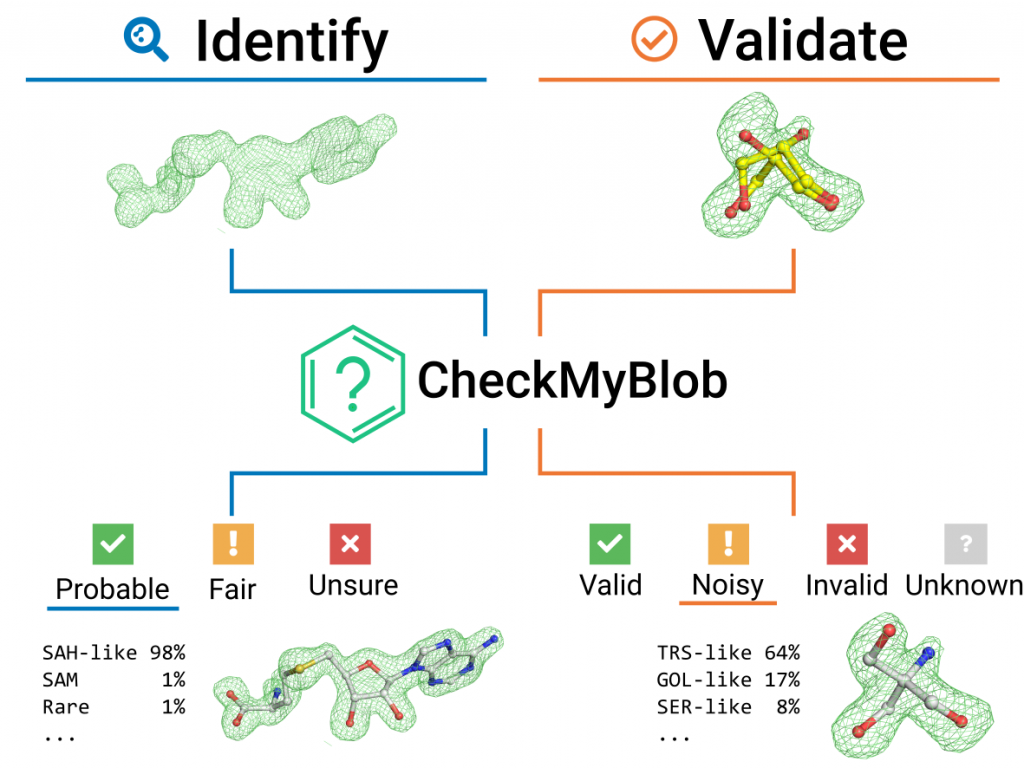
Badania prowadzone przez polsko-amerykański zespół badawczy kierowany przez prof. Wladka Minora, w skład którego wchodzą naukowcy z University of Virginia, Politechniki Poznańskiej, Centrum Instytutu Chemii Bioorganicznej PAN, a także doktorantka realizująca swój projekt badawczy na naszym Wydziale, zaowocowały utworzeniem serwera do automatycznego rozpoznawania małych związków chemicznych w gęstościach elektronowych kryształów białkowych. Wykorzystując uczenie maszynowe naukowcy stworzyli narzędzie, które wspomoże innych badaczy w identyfikacji ligandów w kompleksach makromolekularnych, co może być kluczowe w przypadku projektowania leków opartym na strukturze kompleksu. Serwer przetwarza pliki PDB / mmCIF i MTZ i zwraca ranking 10 najbardziej prawdopodobnych ligandów dla każdej wykrytej chmury gęstości elektronowej wraz z interaktywnymi wizualizacjami 3D. Dodatkowo dla każdej prognozy/walidacji generowany jest skrypt, który umożliwia użytkownikom przeprowadzenie szczegółowej analizy wyników serwera w programie Coot. Serwer CheckMyBlob jest dostępny pod adresem https://checkmyblob.bioreproducibility.org.
Projekt ten był współfinansowany przez Narodową Agencję Wymiany Akademickiej [PPN/BEK/2018/1/00058/U/00001] oraz National Institute of General Medical Sciences [R01-GM132595]. Udział doktorantki, Pani mgr Joanny Macnar, w tym projekcie był możliwy dzięki wsparciu finansowemu uzyskanemu z Programu zintegrowanych działań na rzecz rozwoju Uniwersytetu Warszawskiego (ZIP), współfinansowanego ze środków Europejskiego Funduszu Społecznego w ramach Programu Operacyjnego Wiedza Edukacja Rozwój 2014-2020, ścieżka 3.5.
Dariusz Brzezinski, Przemyslaw J Porebski, Marcin Kowiel, Joanna M Macnar, Wladek Minor, Recognizing and validating ligands with CheckMyBlob, Nucleic Acids Research 49, W1, 2021; https://doi.org/10.1093/nar/gkab296